
Tutorial: particionamento de dados no formato HATS e cross-matching com a biblioteca LSDB¶
Passo-a-passo para conversão de catálogos astronômicos para o formato HATS e execução de cross-matching utilizando a biblioteca LSDB.
Contatos: Luigi Silva (luigi.silva@linea.org.br); Julia Gschwend (julia@linea.org.br).
Última verificação: 27/08/2024
Reconhecimentos¶
'Este notebook utilizou recursos computacionais da Associação Laboratório Interinstitucional de e-Astronomia (LIneA) com o apoio financeiro do INCT do e-Universo (Processo n.º 465376/2014-2).'
'Este notebook se baseia nas bibliotecas e documentações do projeto LSST Interdisciplinary Network for Collaboration and Computing (LINCC) Frameworks, principalmente as bibliotecas hats, hats_import e lsdb. O projeto LINCC Frameworks é apoiado pelo Schmidt Sciences. Ele também é baseado em trabalhos apoiados pela National Science Foundation sob o Subsídio nº AST-2003196. Além disso, ele recebe apoio do DIRAC Institute do Departamento de Astronomia da Universidade de Washington. O DIRAC Institute é apoiado por meio de doações do Charles and Lisa Simonyi Fund for Arts and Sciences, e pelo Washington Research Foundation.'
Introdução¶
Links de referência das bibliotecas de interesse principal¶
Muitos dos textos contidos neste notebook foram extraídos, ou baseados, nos textos das documentações e repositórios das bibliotecas de interesse principal (hats, hats_import e lsdb). A seguir, temos os links para os repositórios e documentações destas bibliotecas.
Repositórios¶
lsdb: https://github.com/astronomy-commons/lsdb
hats_import: https://github.com/astronomy-commons/hats-import
hats: https://github.com/astronomy-commons/hats
Documentações¶
lsdb: https://lsdb.readthedocs.io/en/stable/
hats_import: https://hats-import.readthedocs.io/en/stable/
hats: https://hats.readthedocs.io/en/stable/
HEALPix¶
As bibliotecas hats, hats_import e lsdb utilizam o conceito do HEALPix.
"HEALPix é um acrônimo para Hierarchical Equal Area isoLatitude Pixelization de uma esfera. Como sugerido no nome, essa pixelização produz uma subdivisão de uma superfície esférica na qual cada pixel cobre a mesma área de superfície que todos os outros pixels. A figura abaixo mostra a partição de uma esfera em resoluções progressivamente mais altas, da esquerda para a direita. A esfera verde representa a menor resolução possível com a partição base do HEALPix da superfície esférica em 12 pixels de tamanho igual. A esfera amarela tem uma grade HEALPix de 48 pixels, a esfera vermelha tem 192 pixels e a esfera azul tem uma grade de 768 pixels (resolução de ~7,3 graus).
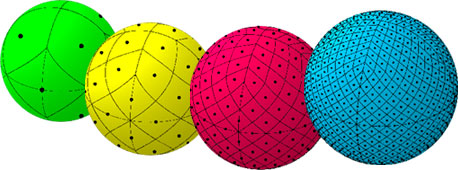
Outra propriedade da grade HEALPix é que os centros dos pixels, representados pelos pontos pretos, ocorrem em um número discreto de anéis de latitude constante. O número de anéis de latitude constante depende da resolução da grade HEALPix. Para as esferas verde, amarela, vermelha e azul mostradas, há 3, 7, 15 e 31 anéis de latitude constante, respectivamente." (HEALPix - NASA)
HATS¶
"Um catálogo HATS (Hierarchical Adaptive Tiling Scheme) é uma partição de objetos em uma esfera. Seu propósito é armazenar dados de grandes levantamentos astronômicos, mas provavelmente poderia ser usado para outros casos de uso onde se tenha grandes volumes de dados com algumas propriedades esféricas." (HATS - GitHub)
Esquema de Particionamento¶
"Nos catálogos no formato HATS, é utilizado o HEALPix (Hierarchical Equal Area isoLatitude Pixelization) para a pixelização esférica e as partições são dimensionadas de forma adaptativa com base no número de objetos.
Em áreas do céu com mais objetos, são usados pixels menores, de modo que todos os pixels resultantes contenham contagens similares de objetos (dentro de uma ordem de magnitude)." (HATS - Docs)
Estrutura de Arquivos¶
"O leitor do catálogo espera encontrar arquivos de acordo com a seguinte estrutura particionada: " (HATS - Docs)
_ /path/to/catalogs/<catalog_name>/
|__ partition_info.csv
|__ properties
|__ dataset/
|__ _common_metadata
|__ _metadata
|__ Norder=1/
| |__ Dir=0/
| |__ Npix=0.parquet
| |__ Npix=1.parquet
|__ Norder=J/
|__ Dir=10000/
|__ Npix=K.parquet
|__ Npix=M.parquet
Cross-matching espacial¶
O cruzamento espacial, ou cross-matching espacial, entre diferentes catálogos astronômicos consiste em identificar e comparar objetos astronômicos de uma mesma região do céu, porém provenientes de diferentes observações.
O cross-matching espacial entre diferentes catalógos é muito útil. Um exemplo desta utilidade pode ser encontrado na elaboração de conjuntos de treinamento para algoritmos de machine learning que calculam redshifts fotométricos. Esses conjuntos de treinamento podem ser elaborados a partir do cruzamento de objetos de um catálogo fotométrico (de onde serão extraídos os features) com objetos de um catálogo espectroscópico (de onde serão extraídos os nossos true redshifts).
Escalabilidade¶
Com o grande volume de dados dos levantamentos atuais e futuros, como o Rubin Observatory Legacy Survey of Space and Time (LSST), o armazenamento e manipulação destes dados é um grande desafio. Catálogos com bilhões de objetos e tamanho de vários terabytes são desafiadores de armazenar e manipular porque exigem hardware de última geração. Processá-los é caro, tanto em termos de tempo de execução quanto de consumo de memória, e realizá-lo em uma única máquina tornou-se impraticável. (LSDB - Docs)
A biblioteca LSDB (Large Survey DataBase) é uma solução que permite a execução escalável de algoritmos. Ele lida com carregamento, consulta, filtragem e cruzamento de dados astronômicos (no formato HATS) em um ambiente distribuído. O framework que permite essa escalabilidade, utilizado pelo LSDB, é o Dask, que aproveita as capacidades de computação distribuída. Com Dask, as operações definidas em um fluxo de trabalho são adicionadas a um gráfico de tarefas que otimiza sua ordem de execução. As operações não são imediatamente computadas - somos nós que decidimos quando iremos computá-las. Assim que iniciamos as computações, o Dask distribui a carga de trabalho entre seus vários dask workers, distribuídos entre os nós de um Cluster, por exemplo, e as tarefas são executadas de maneira eficiente em paralelo. (LSDB - Docs)
Objetos nas bordas¶
A biblioteca LSDB usa os catálogos no formato HATS como forma de organizar os dados espacialmente para, dentre outras coisas, conseguir carregar todos os pontos vizinhos de maneira simultânea, o que é essencial para comparações precisas. No entanto, há uma limitação: nas bordas de cada pixel, alguns pontos serão perdidos. Isso significa que, para operações que exigem comparações com pontos vizinhos, como o cruzamento de dados, o processo pode perder algumas correspondências para pontos próximos às bordas das partições, porque nem todos os pontos próximos são incluídos ao analisar uma partição de cada vez. (LSDB Docs)
Para resolver isso, poderíamos tentar carregar também as partições vizinhas para cada partição que cruzarmos. No entanto, isso significaria carregar muitos dados desnecessários, o que desaceleraria as operações e causaria problemas de falta de memória. Então, para cada catálogo, também criamos um cache de margem. Isso significa que, para cada partição, criamos um arquivo que contém os pontos no catálogo dentro de uma certa distância da borda do pixel. (LSDB Docs)

Importação das bibliotecas e configurações¶
Antes da instalação das bibiliotecas necessárias para esse notebook, é recomendado criar um ambiente virtual. Para isso, você pode seguir os passos contidos na documentação da biblioteca LSDB ou os passos do notebook de tutorial 3-conda-env.ipynb contido no repositório de tutorial do LIneA. Após isso, para utilizar esse ambiente virtual como um kernel no Jupyter Notebook, são necessários os passos a seguir:
conda install -c anaconda ipykernel
python -m ipykernel install --user --name=NOME-DO-SEU-AMBIENTE-VIRTUAL
Estes comandos vão fazer com que o ambiente criado seja disponibilizado como um kernel para o Jupyter Notebook.
As instruções de instalação para as bibliotecas de interesse principal podem ser encontradas nas documentações do LSDB e do HATS Import.
Requisitos para este notebook:
Bibliotecas gerais: os, sys, math, numpy, time, pathlib.
Bibliotecas astronômicas: astropy.
Bibliotecas de computação paralela: dask.
Bibliotecas de visualização: bokeh, holoviews, geoviews, datashader, matplotlib.
Bibliotecas de interesse principal: hats, hats_import, lsdb.
Bibliotecas de manipulação de dados: pandas.
Bibliotecas para a obtenção de dados: dblinea.
Arquivo auxiliar: des-round19-poly.txt (contorno da área coberta pelo levantamento do DES DR2, i.e., DES footprint, 2019 version).
Precisamos fazer o download do arquivo des-round19-poly.txt do repositório kadrlica/skymap no GitHub.
! wget https://raw.githubusercontent.com/kadrlica/skymap/master/skymap/data/des-round19-poly.txt -O des-round19-poly.txt
--2024-10-21 17:07:26-- https://raw.githubusercontent.com/kadrlica/skymap/master/skymap/data/des-round19-poly.txt Resolving raw.githubusercontent.com (raw.githubusercontent.com)... 185.199.110.133, 185.199.111.133, 185.199.109.133, ... Connecting to raw.githubusercontent.com (raw.githubusercontent.com)|185.199.110.133|:443... connected. HTTP request sent, awaiting response... 200 OK Length: 9947 (9.7K) [text/plain] Saving to: ‘des-round19-poly.txt’ des-round19-poly.tx 100%[===================>] 9.71K --.-KB/s in 0s 2024-10-21 17:07:27 (30.1 MB/s) - ‘des-round19-poly.txt’ saved [9947/9947]
Importações¶
Vamos importar as bibliotecas necessárias.
###################### GENERAL ######################
import os
import sys
import math
import numpy as np
import time
from pathlib import Path
from IPython.display import IFrame
###################### ASTRONOMY ######################
### ASTROPY
from astropy.coordinates import SkyCoord
from astropy import units as u
###################### PARALLEL COMPUTING ######################
### DASK
from dask.distributed import Client
###################### VISUALIZATION ######################
### BOKEH
import bokeh
from bokeh.io import output_notebook, show
from bokeh.models import ColorBar, LinearColorMapper
from bokeh.palettes import Viridis256
### HOLOVIEWS
import holoviews as hv
from holoviews import opts
from holoviews.operation.datashader import rasterize, dynspread
### GEOVIEWS
import geoviews as gv
import geoviews.feature as gf
from cartopy import crs
### MATPLOTLIB
import matplotlib.pyplot as plt
###################### HATS AND LSDB ######################
### HATS
## Explore the HATS catalogs and plot sky maps
import hats
from hats.catalog import Catalog
from hats.inspection import plot_pixels
## For converting the data to HATS format and generate margin caches
import hats_import
from hats_import.catalog.file_readers import CsvReader
from hats_import.margin_cache.margin_cache_arguments import MarginCacheArguments
from hats_import.pipeline import ImportArguments, pipeline_with_client
### LDSB
import lsdb
###################### DATA MANAGEMENT ######################
### PANDAS
import pandas as pd
###################### DATA ACCESS ######################
### DB LIneA
from dblinea import DBBase
A seguir, são impressas as versões do Python, Numpy, Bokeh e Holoviews:
print('Python version: ' + sys.version)
print('Numpy version: ' + np.__version__)
print('Bokeh version: ' + bokeh.__version__)
print('HoloViews version: ' + hv.__version__)
print('hats_import version: ' + hats_import.__version__)
print('lsdb version: ' + lsdb.__version__)
Python version: 3.11.9 | packaged by conda-forge | (main, Apr 19 2024, 18:36:13) [GCC 12.3.0] Numpy version: 1.26.4 Bokeh version: 3.4.2 HoloViews version: 1.19.1 hats_import version: 0.4.1.dev2+gaeb92ae lsdb version: 0.4.0
Configurações¶
Definindo o cliente Dask.
client = Client()
Definindo o número de linhas que o pandas irá exibir.
pd.set_option('display.max_rows', 10)
Configurando o holoviews e o geoviews para trabalhar com o bokeh:
hv.extension('bokeh')
gv.extension('bokeh')
Configurando os plots do bokeh para serem em linha:
Configurando os plots do matplotlib para serem em linha:
%matplotlib inline
Leitura dos dados do footprint do DES¶
A seguir, vamos ler o footprint do DES DR2 do arquivo des-round19-poly.txt e imprimir os mínimos e máximos de R.A. e DEC.
foot_ra, foot_dec = np.loadtxt('des-round19-poly.txt', unpack=True)
print("R.A. AND DEC COORDINATES, BEFORE USING SKYCOORD")
print(f"R.A. min: {foot_ra.min():.2f} | R.A. max: {foot_ra.max():.2f}")
print(f"DEC min: {foot_dec.min():.2f} | DEC max: {foot_dec.max():.2f}")
R.A. AND DEC COORDINATES, BEFORE USING SKYCOORD R.A. min: -60.00 | R.A. max: 99.00 DEC min: -66.90 | DEC max: 5.00
Depois de ler o footprint, definimos a classe SkyCoord da biblioteca Astropy usando as coordenadas R.A. e DEC do footprint. Com o SkyCoord, temos uma interface flexível para representação, manipulação e transformação de coordenadas celestes entre sistemas. Usamos também o módulo de unidades do Astropy; em u.degree, por exemplo, indicamos que as coordenadas estão em graus. Além disso, usamos o método wrap_at para garantir que as coordenadas estejam no intervalo $[-180,180)$.
foot_coords = SkyCoord(ra=foot_ra*u.degree, dec=foot_dec*u.degree, frame='icrs')
foot_df = pd.DataFrame({'foot_ra': np.array(foot_coords.ra.wrap_at(180*u.degree)),
'foot_dec': np.array(foot_coords.dec)})
print("R.A. AND DEC COORDINATES, AFTER USING SKYCOORD")
print(f"R.A. min: {foot_df['foot_ra'].min():.2f} | R.A. max: {foot_df['foot_ra'].max():.2f}")
print(f"DEC min: {foot_df['foot_dec'].min():.2f} | DEC max: {foot_df['foot_dec'].max():.2f}")
R.A. AND DEC COORDINATES, AFTER USING SKYCOORD R.A. min: -60.00 | R.A. max: 99.00 DEC min: -66.90 | DEC max: 5.00
Função para gerar os gráficos de distribuição espacial¶
A seguir, temos uma função que retorna os gráficos de distribuição espacial, dadas as coordenadas R.A. e DEC dos objetos. Essa função será usada posteriormente nos plots.
def plot_spatial_distribution(ra_coords, dec_coords, title, title_to_save,
longitudes_ticks=np.arange(30, 360, 30),
latitudes_ticks=np.arange(-75, 76, 15),
height=500, width=1000, padding=0.05,
xlabel='R.A.', ylabel='DEC',
show_des_footprint=True,
show_grid=True,
show_labels=True):
'''Função para exibir os gráficos de distribuição espacial de objetos.
Argumentos:
ra_coords (pandas.core.series.Series, numpy.ndarray ou list): coordenadas R.A. do objeto, em graus.
dec_coords (pandas.core.series.Series, numpy.ndarray ou list): coordenadas DEC do objeto, em graus.
title (str): título a ser exibido no gráfico.
longitudes_ticks (numpy.ndarray): ticks de longitude a serem exibidos no gráfico. PADRÃO: np.arange(30, 360, 30)
latitudes_ticks (numpy.ndarray): ticks de latitude a serem exibidos no gráfico. PADRÃO: np.arange(-75, 76, 15)
height (float): parâmetro relacionado às dimensões do plot. PADRÃO: 500
width (float): parâmetro relacionado às dimensões do plot. PADRÃO: 1000
padding (float): parâmetro relacionado ao posicionamento do plot. PADRÃO: 0.05
xlabel (str): nome do eixo-x. PADRÃO: 'R.A.'
ylabel (str): nome do eixo-y. PADRÃO: 'DEC'
show_des_footprint (boolean): se mostra ou não o footprint do DES. PADRÃO: True
show_grid (boolean): se mostra ou não as linhas de grid. PADRÃO: True
show_labels (boolean): se mostra ou não os ticks de latitude e longitude. PADRÃO: True
'''
### Definindo os ticks de R.A. e DEC. para os plots.
longitudes = longitudes_ticks
latitudes = latitudes_ticks
lon_labels = [f"{lon}°" for lon in longitudes]
lat_labels = [f"{lat}°" for lat in latitudes]
labels_data = {
"lon": list(np.flip(longitudes)) + [-180] * len(latitudes),
"lat": [0] * len(longitudes) + list(latitudes),
"label": lon_labels + lat_labels,
}
df_labels = pd.DataFrame(labels_data)
labels_plot = gv.Labels(df_labels, kdims=["lon", "lat"], vdims=["label"]).opts(
text_font_size="12pt",
text_color="black",
text_align='right',
text_baseline='bottom',
projection=crs.Mollweide()
)
### Definindo as linhas de grid.
grid = gf.grid()
### Definindo a curva do footprint do DES.
### Aqui, multiplicamos as coordenadas 'ra' por (-1) para que o plot fique invertido em R.A. Essa é uma convenção muito utilizada.
ra_dec_foot = gv.Path(((-1)*foot_df['foot_ra'], foot_df['foot_dec'])).opts(line_width=3, color='orange')
### Definindo a classe SkyCoord do astropy para manipulação das coordenadas astronômicas.
ra_dec_coords = SkyCoord(ra=np.array(ra_coords)*u.degree, dec=np.array(dec_coords)*u.degree, frame='icrs')
### Usando o método wrap_at para garantir que as coordenadas estejam no intervalo [-180, 180).
ra_dec_coords = pd.DataFrame({'ra_coords': np.array(ra_dec_coords.ra.wrap_at(180*u.degree)),
'dec_coords': np.array(ra_dec_coords.dec)})
### Definindo o objeto points do geoviews.
### Novamente, multiplicamos as coordenadas 'ra' por (-1) para que o plot fique invertido em R.A. Essa é uma convenção muito utilizada.
ra_dec_points = gv.Points(((-1)*ra_dec_coords['ra_coords'], ra_dec_coords['dec_coords']), kdims=['ra', 'dec'])
### Aplicando a projeção Mollweide.
projected = gv.operation.project(ra_dec_points, projection=crs.Mollweide())
### Aplicando o datashader sobre os pontos.
dsh_points = dynspread(rasterize(projected).opts(cmap="Viridis", cnorm='log'))
dsh_points = dsh_points.opts(width=width, height=height, padding=padding, title=title, toolbar='above', colorbar=True, tools=['box_select'])
### Exibindo o plot.
if show_des_footprint==True:
final_plot = dsh_points * ra_dec_foot * grid * labels_plot
return(hv.save(final_plot, title_to_save))
elif show_grid==True:
final_plot = dsh_points * grid * labels_plot
return(hv.save(final_plot, title_to_save))
elif show_labels==True:
final_plot = dsh_points * labels_plot
return(hv.save(final_plot, title_to_save))
else:
final_plot = dsh_points
return(hv.save(final_plot, title_to_save))
Caracterização da amostra espectroscópica de exemplo¶
Para executar o cross-matching posteriormente, usaremos como dados espectroscópicos uma amostra de objetos do 2dFLenS.
| Nome do levantamento (link para o website) |
Número de redshifts na amostra original |
Referência (link para o artigo) |
|---|---|---|
| 2dFLenS | 70,079 | Blake et al. 2016 |
A amostra que será utilizada será extraída de uma tabela chamada public_specz_compilation via biblioteca DBLIneA. Essa tabela contém um compilado de catálogos de diferentes levantamentos, os quais foram coletados ao longo dos anos de operação do Dark Energy Survey (DES) e agrupados sistematicamente pela ferramenta DES Science Portal (pipeline Spectroscopic Sample) para formar a base de um conjunto de treinamento para algoritmos de cálculo de redshifts fotométricos baseados em machine learning. Temos, no total, dados de 28 levantamentos, como 2DF, 2dFLenS, 3DHST, 6DF, etc.
A seguir, podemos ver as 5 primeiras linhas desse compilado de redshifts fotométricos, a estatística básica dos dados e um plot simples de sua distribuição espacial.
db = DBBase()
schema = 'des_dr2'
tablename = 'public_specz_compilation'
query = f'SELECT * FROM {schema}.{tablename}'
specz_compilation_data = db.fetchall_df(query)
specz_compilation_data.head()
| ra | dec | z | err_z | flag_des | survey | flag_survey | id_spec | |
|---|---|---|---|---|---|---|---|---|
| 0 | 344.762375 | -30.936083 | 0.1141 | 99.0 | 4 | 2DF | 4.0 | 9999 |
| 1 | 353.433375 | -18.816306 | 0.0950 | 99.0 | 4 | 2DF | 5.0 | 9999 |
| 2 | 348.495625 | -34.821056 | 0.2024 | 99.0 | 4 | 2DF | 4.0 | 9999 |
| 3 | 348.505542 | -34.805139 | 0.1424 | 99.0 | 4 | 2DF | 4.0 | 9999 |
| 4 | 348.339167 | -34.702556 | 0.1426 | 99.0 | 4 | 2DF | 4.0 | 9999 |
specz_compilation_data.describe()
| ra | dec | z | err_z | flag_des | flag_survey | id_spec | |
|---|---|---|---|---|---|---|---|
| count | 3.661690e+06 | 3.661690e+06 | 3.661690e+06 | 3.661690e+06 | 3.661690e+06 | 3.661690e+06 | 3661690.0 |
| mean | 1.700340e+02 | 1.592614e+01 | 4.642166e-01 | 9.451649e+01 | 3.917151e+00 | 3.844424e+00 | 9999.0 |
| std | 9.680731e+01 | 2.456493e+01 | 4.547598e-01 | 2.073263e+01 | 2.756537e-01 | 7.167758e-01 | 0.0 |
| min | 9.458086e-05 | -8.713647e+01 | 6.671280e-06 | -9.999000e+00 | 3.000000e+00 | 0.000000e+00 | 9999.0 |
| 25% | 1.239519e+02 | 1.174380e-01 | 1.461030e-01 | 9.900000e+01 | 4.000000e+00 | 4.000000e+00 | 9999.0 |
| 50% | 1.735271e+02 | 1.455978e+01 | 4.316670e-01 | 9.900000e+01 | 4.000000e+00 | 4.000000e+00 | 9999.0 |
| 75% | 2.223870e+02 | 3.359415e+01 | 6.102090e-01 | 9.900000e+01 | 4.000000e+00 | 4.000000e+00 | 9999.0 |
| max | 3.599998e+02 | 8.427039e+01 | 7.010940e+00 | 4.845040e+03 | 4.000000e+00 | 2.900000e+01 | 9999.0 |
ra_coords = specz_compilation_data['ra']
dec_coords = specz_compilation_data['dec']
title = 'Distribuição espacial - Todos os objetos contidos na tabela Public spec-z compilation'
title_to_save = 'all_public_specz_compilation_objects.html'
plot_spatial_distribution(ra_coords, dec_coords, title, title_to_save)
IFrame('all_public_specz_compilation_objects.html', width=1200, height=600)
Observação: pode acontecer um bug de reenderização nos plots que subestime/superestime os objetos por pixel. Um simples clique na ferramenta "reset", na barra de ferramentas do Bokeh na parte superior do plot, deve resolver o problema. Se não resolver, certifique-se de que todos os pacotes e extensões foram instaladas corretamente, de forma que os gráficos estejam dinâmicos (reenderizam conforme o zoom dado). Em caso de dúvida, verifique o arquivo de instruções instructions_hispcat_lsdb_tutorial.md, que está no mesmo diretório deste notebook.
Além de combinar todos os catálogos em uma única tabela, o pipeline Spectroscopic Sample também homogeneiza as várias flags de qualidade originais dos catálogos em um único sistema (flag_des) baseado nos parâmetros usados no levantamento OzDES (Yuan et al., 2015). Em resumo, as flags significam:
| flag_des | Significado |
|---|---|
| 1 | redshift desconhecido |
| 2 | palpite não confiável |
| 3 | 95% de confiança |
| 4 | 99% de confiança |
Como a tabela public_specz_compilation contém um compilado de redshifts espectroscópicos de vários levantamentos astronômicos, muitos desses levantamentos observaram regiões comuns do céu. Ao agrupar todas as medidas em um catálogo único, geralmente há medidas múltiplas de redshift espectroscópico para um mesmo objeto. Para identificar esses casos, o pipeline Spectroscopic Sample fez uma combinação espacial entre as coordenadas equatoriais de "todos contra todos" com um raio de busca de 1.0 arcsec de cada objeto. Então, ele aplicou uma seleção para manter apenas uma medida para cada objeto extragaláctico presente na amostra, seguindo o critério abaixo para escolha e desempate:
- medida com a maior flag de qualidade (
flag_des) - medida com o menor erro no redshift (
err_z) - medida obtida pelo levantamento mais recente
Um corte de qualidade também foi aplicado onde apenas objetos com flag_des ⩾ 3 foram incluídos no compilado.
Portanto, como a amostra do 2dFLenS que usaremos será um subconjunto dos dados contidos na tabela public spec-z compilation, estamos pegando, nessa amostra, apenas aqueles objetos que permaneceram após a seleção considerando todos os outros catálogos presentes no compilado de redshifts fotométricos, e apenas os objetos com flag_des ⩾ 3.
Todos os objetos do 2dFLenS, contidos no produto Public spec-z compilation¶
Primeiramente, vamos filtrar os dados do compilado para obter apenas aqueles referentes ao 2dFLenS.
df_specz_sample = specz_compilation_data[specz_compilation_data['survey']=='2dFLenS']
A seguir, temos as primeiras 5 linhas da tabela contendo a amostra.
df_specz_sample.head()
| ra | dec | z | err_z | flag_des | survey | flag_survey | id_spec | |
|---|---|---|---|---|---|---|---|---|
| 188825 | 169.990925 | -6.246967 | 0.48565 | 99.0 | 3 | 2dFLenS | 3.0 | 9999 |
| 188826 | 178.058477 | -4.655283 | 0.57433 | 99.0 | 4 | 2dFLenS | 4.0 | 9999 |
| 188827 | 171.796769 | -7.245199 | 0.65118 | 99.0 | 3 | 2dFLenS | 3.0 | 9999 |
| 188828 | 175.656537 | -4.328665 | 0.27805 | 99.0 | 3 | 2dFLenS | 3.0 | 9999 |
| 188829 | 171.687131 | -7.061002 | 0.59698 | 99.0 | 4 | 2dFLenS | 4.0 | 9999 |
Temos também as estatísticas básicas dos dados.
df_specz_sample.describe()
| ra | dec | z | err_z | flag_des | flag_survey | id_spec | |
|---|---|---|---|---|---|---|---|
| count | 63240.000000 | 63240.000000 | 63240.000000 | 63240.0 | 63240.000000 | 63240.000000 | 63240.0 |
| mean | 164.605564 | -23.708983 | 0.343966 | 99.0 | 3.730487 | 3.782005 | 9999.0 |
| std | 136.720066 | 11.630184 | 0.240018 | 0.0 | 0.443710 | 0.570116 | 0.0 |
| min | 0.002231 | -35.747117 | 0.001000 | 99.0 | 3.000000 | 3.000000 | 9999.0 |
| 25% | 30.338937 | -31.928488 | 0.166220 | 99.0 | 3.000000 | 3.000000 | 9999.0 |
| 50% | 165.625466 | -29.353255 | 0.293250 | 99.0 | 4.000000 | 4.000000 | 9999.0 |
| 75% | 335.988548 | -8.938086 | 0.515563 | 99.0 | 4.000000 | 4.000000 | 9999.0 |
| max | 359.993683 | -0.957408 | 4.688020 | 99.0 | 4.000000 | 6.000000 | 9999.0 |
Em seguida, fazemos o plot de distribuição de objetos.
ra_coords = df_specz_sample['ra']
dec_coords = df_specz_sample['dec']
title = 'Distribuição espacial - Todos os objetos do 2dFLenS, contidos na tabela Public spec-z compilation'
title_to_save = 'all_2df_lens_objects_in_the_public_specz_compilation.html'
plot_spatial_distribution(ra_coords, dec_coords, title, title_to_save)
IFrame('all_2df_lens_objects_in_the_public_specz_compilation.html', width=1200, height=600)
Observação: pode acontecer um bug de reenderização nos plots que subestime/superestime os objetos por pixel. Um simples clique na ferramenta "reset", na barra de ferramentas do Bokeh na parte superior do plot, deve resolver o problema. Se não resolver, certifique-se de que todos os pacotes e extensões foram instaladas corretamente, de forma que os gráficos estejam dinâmicos (reenderizam conforme o zoom dado). Em caso de dúvida, verifique o arquivo de instruções instructions_hispcat_lsdb_tutorial.md, que está no mesmo diretório deste notebook.
Objetos do 2dFLenS filtrados para pertencerem ao footprint do DES¶
A seguir, faremos um filtro simples para obter os dados do 2dFLenS dentro do footprint do DES. O filtro a seguir irá guardar objetos com $ra<90$ ou $ra>359$, e $dec < -15$.
df_specz_sample_filtered = df_specz_sample.query("ra > 359 | ra < 90. & dec < -15.")
Mostrando as primeiras 5 linhas da tabela contendo a amostra filtrada.
df_specz_sample_filtered.head()
| ra | dec | z | err_z | flag_des | survey | flag_survey | id_spec | |
|---|---|---|---|---|---|---|---|---|
| 193047 | 359.764587 | -31.877622 | 0.11551 | 99.0 | 4 | 2dFLenS | 4.0 | 9999 |
| 193288 | 359.471130 | -31.757070 | 0.06057 | 99.0 | 4 | 2dFLenS | 4.0 | 9999 |
| 193434 | 359.456970 | -34.257616 | 0.39392 | 99.0 | 3 | 2dFLenS | 3.0 | 9999 |
| 193443 | 359.510864 | -34.252725 | 0.11191 | 99.0 | 4 | 2dFLenS | 4.0 | 9999 |
| 193450 | 359.485443 | -34.226701 | 0.11504 | 99.0 | 4 | 2dFLenS | 4.0 | 9999 |
Estatística básica desses dados filtrados.
df_specz_sample_filtered.describe()
| ra | dec | z | err_z | flag_des | flag_survey | id_spec | |
|---|---|---|---|---|---|---|---|
| count | 26652.000000 | 26652.000000 | 26652.000000 | 26652.0 | 26652.000000 | 26652.000000 | 26652.0 |
| mean | 33.268846 | -30.771159 | 0.342813 | 99.0 | 3.753489 | 3.754465 | 9999.0 |
| std | 53.858067 | 2.024508 | 0.248144 | 0.0 | 0.430987 | 0.433799 | 0.0 |
| min | 0.002231 | -35.723279 | 0.001010 | 99.0 | 3.000000 | 3.000000 | 9999.0 |
| 25% | 11.858320 | -32.484795 | 0.166837 | 99.0 | 4.000000 | 4.000000 | 9999.0 |
| 50% | 25.023028 | -30.907806 | 0.284645 | 99.0 | 4.000000 | 4.000000 | 9999.0 |
| 75% | 39.911329 | -29.043815 | 0.504690 | 99.0 | 4.000000 | 4.000000 | 9999.0 |
| max | 359.993683 | -26.253114 | 4.688020 | 99.0 | 4.000000 | 6.000000 | 9999.0 |
Em seguida, fazemos o plot de distribuição de objetos.
ra_coords = df_specz_sample_filtered['ra']
dec_coords = df_specz_sample_filtered['dec']
title = 'Distribuição espacial - Objetos do 2dFLenS filtrados para pertencerem ao footprint do DES'
title_to_save = '2dflens_objects_filtered_to_be_in_DES_footprint.html'
plot_spatial_distribution(ra_coords, dec_coords, title, title_to_save)
IFrame('2dflens_objects_filtered_to_be_in_DES_footprint.html', width=1200, height=600)
Observação: pode acontecer um bug de reenderização nos plots que subestime/superestime os objetos por pixel. Um simples clique na ferramenta "reset", na barra de ferramentas do Bokeh na parte superior do plot, deve resolver o problema. Se não resolver, certifique-se de que todos os pacotes e extensões foram instaladas corretamente, de forma que os gráficos estejam dinâmicos (reenderizam conforme o zoom dado). Em caso de dúvida, verifique o arquivo de instruções instructions_hispcat_lsdb_tutorial.md, que está no mesmo diretório deste notebook.
Por fim, salvamos os dados em um arquivo. Antes de salvar, no entanto, resetamos os índices para que eles sejam inteiros sequenciais, para que possamos executar o cross-matching posteriormente. Os índices originais da tabela serão salvos na coluna "index".
! mkdir -p data
! mkdir -p data/input
! mkdir -p data/input/specz
### Resetando os índices.
df_specz_sample_filtered_reseted = df_specz_sample_filtered.reset_index()
df_specz_sample_filtered_reseted.head()
| index | ra | dec | z | err_z | flag_des | survey | flag_survey | id_spec | |
|---|---|---|---|---|---|---|---|---|---|
| 0 | 193047 | 359.764587 | -31.877622 | 0.11551 | 99.0 | 4 | 2dFLenS | 4.0 | 9999 |
| 1 | 193288 | 359.471130 | -31.757070 | 0.06057 | 99.0 | 4 | 2dFLenS | 4.0 | 9999 |
| 2 | 193434 | 359.456970 | -34.257616 | 0.39392 | 99.0 | 3 | 2dFLenS | 3.0 | 9999 |
| 3 | 193443 | 359.510864 | -34.252725 | 0.11191 | 99.0 | 4 | 2dFLenS | 4.0 | 9999 |
| 4 | 193450 | 359.485443 | -34.226701 | 0.11504 | 99.0 | 4 | 2dFLenS | 4.0 | 9999 |
### Salvando o arquivo.
df_specz_sample_filtered_reseted.to_parquet('data/input/specz/specz-2dflens-sample.pq', index=False)
Caracterização da amostra fotométrica de exemplo¶
Para executar o cross-matching posteriormente, usaremos como dados fotométricos uma amostra de objetos do DES DR2.
| Nome do levantamento (link para o website) |
Número de objetos na tabela original |
Referência (link para o artigo) |
|---|---|---|
| DES DR2 | ~691 milhões de objetos astronômicos distintos | DES Collaboration 2021 |
Para obter esses dados, utilizaremos a biblioteca dblinea. Aqui, nós vamos acessar os dados da tabela coadd_objects do catálogo DES DR2.
schema = "des_dr2"
tablename = "main"
A tabela original tem 215 colunas. O nome e significado de cada coluna pode ser encontrado aqui. Aqui, iremos utilizar as seguintes colunas:
| Coluna | Significado |
|---|---|
| COADD_OBJECT_ID | Identificador único para os objetos co-adicionados |
| RA | Ascensão reta, com precisão quantizada para indexação (ALPHAWIN_J2000 tem precisão total, mas não indexada) [graus] |
| DEC | Declinação, com precisão quantizada para indexação (DELTAWIN_J2000 tem precisão total, mas não indexada) [graus] |
| MAG_AUTO_{G,R,I,Z,Y}_DERED | Estimativa de magnitude desavermelhada (usando SFD98), para um modelo elíptico baseado no raio de Kron [mag] |
| MAGERR_AUTO_{G,R,I,Z,Y} | Incerteza na estimativa de magnitude, para um modelo elíptico baseado no raio de Kron [mag] |
| FLAGS_{G,R,I,Z,Y} | Flag aditiva que descreve conselhos preventivos sobre o processo de extração da fonte. Use menos de 4 para objetos bem comportados |
| EXTENDED_CLASS_COADD | 0: estrelas de alta confiança; 1: estrelas candidatas; 2: principalmente galáxias; 3: galáxias de alta confiança; -9: Sem dados; Usando fotometria Sextractor |
Além disso, a tabela original tem muitos dados. Seria inviável, em termos computacionais, pegar todos os dados que coincidem com a região do 2dFLenS neste notebook. Portanto, iremos restringir a busca para uma região pequena. Aqui, usaremos a região com $7 < R.A < 10$ e $-33 < DEC < -30$.
### Definindo as coordenadas para a query. Essas variáveis também serão usadas posteriormente nos plots.
ra_min = 7
ra_max = 10
dec_min = -33
dec_max = -30
### Executando a query.
query = (f"SELECT coadd_object_id, ra, dec, mag_auto_g_dered, mag_auto_r_dered, mag_auto_i_dered, mag_auto_z_dered, mag_auto_y_dered, magerr_auto_g, "+
f"magerr_auto_r, magerr_auto_i, magerr_auto_z, magerr_auto_y, flags_g, flags_r, flags_i, flags_z, flags_y, extended_class_coadd "+
f"FROM {schema}.{tablename} "+
f"WHERE (dec <= {dec_max} AND dec >= {dec_min} AND ra <= {ra_max} AND ra>={ra_min}) "
)
%%time
df_photo_sample = db.fetchall_df(query)
CPU times: user 17.4 s, sys: 3.44 s, total: 20.8 s Wall time: 33.9 s
A seguir, temos as primeiras cinco linhas da tabela contendo a amostra.
df_photo_sample.head()
| coadd_object_id | ra | dec | mag_auto_g_dered | mag_auto_r_dered | mag_auto_i_dered | mag_auto_z_dered | mag_auto_y_dered | magerr_auto_g | magerr_auto_r | magerr_auto_i | magerr_auto_z | magerr_auto_y | flags_g | flags_r | flags_i | flags_z | flags_y | extended_class_coadd | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 1068916420 | 7.041218 | -32.998839 | 21.221159 | 20.416883 | 19.983702 | 18.403831 | 18.723942 | 0.076484 | 0.046852 | 0.039773 | 0.030717 | 0.065074 | 3 | 3 | 3 | 3 | 3 | 3 |
| 1 | 1068917101 | 7.055067 | -32.998628 | 22.957352 | 99.000000 | 99.000000 | 20.442957 | 20.263426 | 0.160077 | 99.000000 | 99.000000 | 0.087319 | 0.112613 | 3 | 3 | 3 | 3 | 3 | 1 |
| 2 | 1068917147 | 7.057777 | -32.999975 | 21.614515 | 20.479462 | 19.812565 | 19.401255 | 19.375076 | 0.024625 | 0.011223 | 0.008306 | 0.017520 | 0.027179 | 0 | 0 | 0 | 0 | 0 | 0 |
| 3 | 1068916936 | 7.061899 | -32.997800 | 22.444773 | 21.114576 | 20.274847 | 19.958334 | 19.877291 | 0.055731 | 0.019563 | 0.013175 | 0.031619 | 0.046797 | 0 | 0 | 0 | 0 | 0 | 3 |
| 4 | 1068917057 | 7.063429 | -32.997956 | 24.776548 | 99.000000 | 23.550138 | 22.857664 | 99.000000 | 0.284739 | 99.000000 | 0.175163 | 0.268928 | 99.000000 | 0 | 0 | 0 | 0 | 0 | 2 |
Temos também as statísticas básicas dos dados.
df_photo_sample.describe()
| coadd_object_id | ra | dec | mag_auto_g_dered | mag_auto_r_dered | mag_auto_i_dered | mag_auto_z_dered | mag_auto_y_dered | magerr_auto_g | magerr_auto_r | magerr_auto_i | magerr_auto_z | magerr_auto_y | flags_g | flags_r | flags_i | flags_z | flags_y | extended_class_coadd | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| count | 1.004484e+06 | 1.004484e+06 | 1.004484e+06 | 1.004484e+06 | 1.004484e+06 | 1.004484e+06 | 1.004484e+06 | 1.004484e+06 | 1.004484e+06 | 1.004484e+06 | 1.004484e+06 | 1.004484e+06 | 1.004484e+06 | 1.004484e+06 | 1.004484e+06 | 1.004484e+06 | 1.004484e+06 | 1.004484e+06 | 1.004484e+06 |
| mean | 1.079497e+09 | 8.495268e+00 | -3.148407e+01 | 2.676091e+01 | 2.426884e+01 | 2.370173e+01 | 2.343840e+01 | 3.610558e+01 | 3.534941e+00 | 6.834363e-01 | 5.271135e-01 | 8.130552e-01 | 2.007098e+01 | 4.328601e-01 | 4.280576e-01 | 4.279491e-01 | 4.284598e-01 | 4.607032e-01 | 2.204870e+00 |
| std | 5.668166e+06 | 8.643299e-01 | 8.679997e-01 | 1.250962e+01 | 4.970130e+00 | 3.855900e+00 | 4.586541e+00 | 2.884006e+01 | 5.237295e+01 | 2.697540e+01 | 9.607355e+00 | 3.417773e+01 | 1.724621e+02 | 1.031740e+00 | 9.932178e-01 | 9.918720e-01 | 9.965610e-01 | 1.219350e+00 | 8.353268e-01 |
| min | 1.068863e+09 | 7.000003e+00 | -3.300000e+01 | 1.121649e+01 | 1.065039e+01 | 1.039437e+01 | 9.536492e+00 | 8.077494e+00 | 4.774237e-05 | 4.272211e-05 | 4.113756e-05 | 2.960894e-05 | 2.897646e-05 | 0.000000e+00 | 0.000000e+00 | 0.000000e+00 | 0.000000e+00 | 0.000000e+00 | -9.000000e+00 |
| 25% | 1.074787e+09 | 7.748380e+00 | -3.222693e+01 | 2.395451e+01 | 2.331324e+01 | 2.282399e+01 | 2.246457e+01 | 2.228145e+01 | 1.218456e-01 | 8.939417e-02 | 1.049233e-01 | 1.514463e-01 | 3.720376e-01 | 0.000000e+00 | 0.000000e+00 | 0.000000e+00 | 0.000000e+00 | 0.000000e+00 | 2.000000e+00 |
| 50% | 1.080077e+09 | 8.493845e+00 | -3.147791e+01 | 2.479343e+01 | 2.423810e+01 | 2.383352e+01 | 2.348004e+01 | 2.339712e+01 | 2.139485e-01 | 1.669808e-01 | 2.124021e-01 | 3.038787e-01 | 7.988981e-01 | 0.000000e+00 | 0.000000e+00 | 0.000000e+00 | 0.000000e+00 | 0.000000e+00 | 2.000000e+00 |
| 75% | 1.085106e+09 | 9.243046e+00 | -3.073277e+01 | 2.557208e+01 | 2.494531e+01 | 2.457013e+01 | 2.421742e+01 | 2.497062e+01 | 3.836854e-01 | 2.647245e-01 | 3.381309e-01 | 4.821458e-01 | 3.091160e+00 | 0.000000e+00 | 0.000000e+00 | 0.000000e+00 | 0.000000e+00 | 0.000000e+00 | 3.000000e+00 |
| max | 1.091113e+09 | 1.000000e+01 | -3.000000e+01 | 9.900000e+01 | 9.900000e+01 | 9.900000e+01 | 9.900000e+01 | 9.900000e+01 | 4.098253e+04 | 2.578874e+04 | 5.794771e+03 | 3.116713e+04 | 9.520319e+04 | 2.600000e+01 | 2.600000e+01 | 2.600000e+01 | 2.600000e+01 | 3.000000e+01 | 3.000000e+00 |
A seguir, fazemos um plot da distribuição espacial desses objetos.
ra_coords = df_photo_sample['ra']
dec_coords = df_photo_sample['dec']
title = 'Distribuição espacial - Amostra do DES DR2'
title_to_save = 'sample_of_des_dr2.html'
plot_spatial_distribution(ra_coords, dec_coords, title, title_to_save)
IFrame('sample_of_des_dr2.html', width=1200, height=600)
Observação: pode acontecer um bug de reenderização nos plots que subestime/superestime os objetos por pixel. Um simples clique na ferramenta "reset", na barra de ferramentas do Bokeh na parte superior do plot, deve resolver o problema. Se não resolver, certifique-se de que todos os pacotes e extensões foram instaladas corretamente, de forma que os gráficos estejam dinâmicos (reenderizam conforme o zoom dado). Em caso de dúvida, verifique o arquivo de instruções instructions_hispcat_lsdb_tutorial.md, que está no mesmo diretório deste notebook.
Por fim, salvamos os dados em um arquivo.
! mkdir -p data
! mkdir -p data/input
! mkdir -p data/input/photo
df_photo_sample.to_parquet('data/input/photo/photo-des-dr2-sample.pq', index=False)
Utilizando o hats_import¶
A biblioteca hats_import é um utilitário para converter grandes volumes de dados de levantamentos astronômicos para a estrutura HATS. A seguir, iremos utilizá-la para converter os dados espectroscópicos e fotométricos para o formato HATS.
Instalação do hats_import¶
A biblioteca hats_import normalmente pode ser instalada via pip com o comando:
pip install hats-import
Para mais detalhes sobre a instalação, veja a documentação neste link.
Conversão para o formato HATS dos dados espectroscópicos¶
Para os passos a seguir, foram utilizados, principalmente, os seguintes exemplos de referência:
https://lsdb.readthedocs.io/en/stable/tutorials/pre_executed/des-gaia.html
https://hats-import.readthedocs.io/en/stable/catalogs/public/sdss.html
Primeiramente, vamos ler o arquivo Parquet que salvamos para conferir a estrutura da tabela.
pd.read_parquet("data/input/specz/specz-2dflens-sample.pq").head()
| index | ra | dec | z | err_z | flag_des | survey | flag_survey | id_spec | |
|---|---|---|---|---|---|---|---|---|---|
| 0 | 193047 | 359.764587 | -31.877622 | 0.11551 | 99.0 | 4 | 2dFLenS | 4.0 | 9999 |
| 1 | 193288 | 359.471130 | -31.757070 | 0.06057 | 99.0 | 4 | 2dFLenS | 4.0 | 9999 |
| 2 | 193434 | 359.456970 | -34.257616 | 0.39392 | 99.0 | 3 | 2dFLenS | 3.0 | 9999 |
| 3 | 193443 | 359.510864 | -34.252725 | 0.11191 | 99.0 | 4 | 2dFLenS | 4.0 | 9999 |
| 4 | 193450 | 359.485443 | -34.226701 | 0.11504 | 99.0 | 4 | 2dFLenS | 4.0 | 9999 |
Agora, vamos definir os diretórios para salvar os dados no formato HATS.
! mkdir -p data
! mkdir -p data/hats
### Diretório dos dados espectroscópicos de input.
_2DFLENS_DIR = Path("data/input/specz/")
### Nomes para os diretórios do catalógo e do cache de margem dos dados espectroscópicos no formato HATS.
_2DFLENS_HATS_NAME = "_2dflens"
_2DFLENS_MARGIN_CACHE_NAME = "_2dflens_margin_cache"
### Definindo os diretórios para os arquivos HATS com base nos nomes definidos anteriormente.
HATS_DIR = Path("data/hats/")
_2DFLENS_HATS_DIR = HATS_DIR / _2DFLENS_HATS_NAME
_2DFLENS_MARGIN_CACHE_DIR = HATS_DIR / _2DFLENS_MARGIN_CACHE_NAME
Vamos, então, converter os dados espectroscópicos para o formato HATS. Para isso, precisamos indicar para o pipeline as colunas contendo os IDs dos objetos, as coordenadas R.A. e DEC, o arquivo de input e o tipo deste arquivo (no nosso caso, um arquivo Parquet) e o nome e o diretório do catálogo HATS de output.
Além disso, ao criar um novo catálogo utilizando o hats_import, o pipeline tenta criar partições com aproximadamente o memso número de linhas por partição. Esse processo não é perfeito, porém, mesmo assim, o pipeline tenta criar pixels de área menor em áreas mais densas e pixels de área maior em áreas menos densas.
O argumento pixel_threshold é usado para indicar até que ponto o pipeline deve dividir uma certa partição. Ele irá dividir a partição em pixels HEALPix cada vez menores até que o número de linhas fique menor que o pixel_theshold, parando o processo. O processo também pode parar caso hajam tantas divisões que ultrapassem o parâmetro highest_healpix_order (você pode conferir a ordem máxima padrão neste link). Se for preciso dividir ainda mais, surgirá um erro na etapa "Binning" e os parâmetros devem ser ajustados.
Para mais detalhes, veja a documentação.
Convertendo os dados para o formato hats (DESCOMENTE A ÚLTIMA LINHA PARA RODAR O PIPELINE):
_2dflens_args = ImportArguments(
sort_columns="index",
ra_column="ra",
dec_column="dec",
input_path=_2DFLENS_DIR,
file_reader="parquet",
output_artifact_name=_2DFLENS_HATS_NAME,
output_path=HATS_DIR,
pixel_threshold=1_000,
)
### RUN THE PIPELINE (it will fail if the HATS catalog already exists)
#pipeline_with_client(_2dflens_args, client)
Fazendo o plot dos pixels:
# Read the HATS catalog metadata, it does not load any data, just healpix pixels and other metadata
_2dflens_hats_catalog = Catalog.read_hats(_2DFLENS_HATS_DIR)
plot_pixels(_2dflens_hats_catalog)
Posteriormente, desejamos fazer o cross-matching desses dados espectrocópicos (2dFLenS) no formato HATS com os dados fotométricos (DES DR2).
O cross-matching, executado pela biblioteca LSDB, não é simétrico, o que significa que a escolha de qual catálogo é o "esquerdo" e qual é o "direito" é crucial. No nosso caso, iremos fazer o cross-matching do DES DR2 (esquerdo) com o 2dFLenS (direito). Essa configuração geralmente permite que múltiplos objetos DES sejam correspondidos a um único objeto do 2dFLenS, um resultado dos caches de margem. Os caches de margem são projetados para evitar a perda de objetos próximos às bordas dos tiles HEALPix. No entanto, eles podem levar a múltiplas correspondências, onde o mesmo objeto do 2dFLenS pode corresponder a um objeto DES em uma partição e a outro objeto DES na partição vizinha que inclui esse objeto do 2dFLenS em seu cache de margem.
Portanto, para o cross-matching, a biblioteca LSDB precisa do cache de margem do catálogo direito para gerar o resultado completo do cruzamento. Sem o cache de margem, os objetos localizados perto das bordas dos tiles Healpix podem ser perdidos no cruzamento. Veja mais detalhes neste link.
Gerando o cache de margem para o 2dFLenS (DESCOMENTE A ÚLTIMA LINHA PARA RODAR O PIPELINE):
margin_cache_args = MarginCacheArguments(
input_catalog_path=_2DFLENS_HATS_DIR,
output_path=HATS_DIR,
margin_threshold=5.0, # arcsec
output_artifact_name=_2DFLENS_MARGIN_CACHE_NAME,
)
### RUN THE PIPELINE (it will fail if the margin cache already exists)
#pipeline_with_client(margin_cache_args, client)
Conversão para o formato hats dos dados fotométricos¶
Para os passos a seguir, foram utilizados, principalmente, os seguintes exemplos de referência:
https://lsdb.readthedocs.io/en/stable/tutorials/pre_executed/des-gaia.html
https://hats-import.readthedocs.io/en/stable/catalogs/public/sdss.html
Primeiramente, vamos ler o arquivo Parquet que salvamos para conferir a estrutura da tabela.
pd.read_parquet("data/input/photo/photo-des-dr2-sample.pq").head()
| coadd_object_id | ra | dec | mag_auto_g_dered | mag_auto_r_dered | mag_auto_i_dered | mag_auto_z_dered | mag_auto_y_dered | magerr_auto_g | magerr_auto_r | magerr_auto_i | magerr_auto_z | magerr_auto_y | flags_g | flags_r | flags_i | flags_z | flags_y | extended_class_coadd | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 1068916420 | 7.041218 | -32.998839 | 21.221159 | 20.416883 | 19.983702 | 18.403831 | 18.723942 | 0.076484 | 0.046852 | 0.039773 | 0.030717 | 0.065074 | 3 | 3 | 3 | 3 | 3 | 3 |
| 1 | 1068917101 | 7.055067 | -32.998628 | 22.957352 | 99.000000 | 99.000000 | 20.442957 | 20.263426 | 0.160077 | 99.000000 | 99.000000 | 0.087319 | 0.112613 | 3 | 3 | 3 | 3 | 3 | 1 |
| 2 | 1068917147 | 7.057777 | -32.999975 | 21.614515 | 20.479462 | 19.812565 | 19.401255 | 19.375076 | 0.024625 | 0.011223 | 0.008306 | 0.017520 | 0.027179 | 0 | 0 | 0 | 0 | 0 | 0 |
| 3 | 1068916936 | 7.061899 | -32.997800 | 22.444773 | 21.114576 | 20.274847 | 19.958334 | 19.877291 | 0.055731 | 0.019563 | 0.013175 | 0.031619 | 0.046797 | 0 | 0 | 0 | 0 | 0 | 3 |
| 4 | 1068917057 | 7.063429 | -32.997956 | 24.776548 | 99.000000 | 23.550138 | 22.857664 | 99.000000 | 0.284739 | 99.000000 | 0.175163 | 0.268928 | 99.000000 | 0 | 0 | 0 | 0 | 0 | 2 |
Agora, vamos definir os diretórios para salvar os dados no formato HATS.
! mkdir -p data
! mkdir -p data/hats
### Diretório dos dados fotométricos de input.
DES_DIR = Path("data/input/photo/")
### Nome para o diretório do catalógo dos dados fotométricos no formato HATS.
DES_HATS_NAME = "des_dr2"
### Definindo o diretório para os arquivos HATS com base no nome definido anteriormente.
HATS_DIR = Path("data/hats/")
DES_HATS_DIR = HATS_DIR / DES_HATS_NAME
Da mesma forma que foi feito para os dados espectroscópicos, vamos, a seguir, converter os dados fotométricos para o formato HATS. Como o DES DR2 é o nosso catálogo "esquerdo", não precisamos gerar um cache de margem para ele.
Convertendo os dados para o formato hats (DESCOMENTE A ÚLTIMA LINHA PARA RODAR O PIPELINE):
des_args = ImportArguments(
sort_columns="coadd_object_id",
ra_column="ra",
dec_column="dec",
input_path=DES_DIR,
file_reader="parquet",
output_artifact_name=DES_HATS_NAME,
output_path=HATS_DIR,
pixel_threshold=30_000,
)
### RUN THE PIPELINE (it will fail if the HATS catalog already exists)
#pipeline_with_client(des_args, client)
Fazendo o plot dos pixels:
# Read the HATS catalog metadata, it does not load any data, just healpix pixels and other metadata
des_hats_catalog = Catalog.read_hats(DES_HATS_DIR)
plot_pixels(des_hats_catalog)
Utilizando o LSDB¶
"A biblioteca LSDB é um framework que facilita e permite a análise espacial rápida de catálogos astronômicos extremamente grandes (ou seja, consulta e cruzamento de O(1B) fontes). Ela visa abordar os desafios do processamento de dados em larga escala, em particular aqueles levantados pelo LSST.
Construída sobre o Dask para escalar e paralelizar operações de forma eficiente em vários dask workers, ela aproveita o formato de dados HATS para levantamentos em uma estrutura particionada HEALPix." (LSDB Docs)
Instalação do LSDB¶
A biblioteca LSDB normalmente pode ser instalada via conda ou pip com os comandos:
conda install -c conda-forge lsdb
python -m pip install lsdb
Para mais detalhes sobre a instalação, veja a documentação neste link.
X-matching usando o LSDB¶
Para os passos a seguir, foi utilizado, principalmente, o seguinte exemplo de referência:
https://lsdb.readthedocs.io/en/stable/tutorials/pre_executed/des-gaia.html
Definimos, a seguir, o nome do diretório que armazenará os dados do crossmatch.
XMATCH_NAME = "des_dr2_x_2dflens"
OUTPUT_HATS_DIR = HATS_DIR / XMATCH_NAME
A seguir, lemos os dados espectroscópicos e fotométricos, previamente salvos no formato hats.
des_catalog = lsdb.read_hats(DES_HATS_DIR)
_2dflens_margin_cache_catalog = lsdb.read_hats(_2DFLENS_MARGIN_CACHE_DIR)
_2dflens_catalog = lsdb.read_hats(_2DFLENS_HATS_DIR, margin_cache=_2dflens_margin_cache_catalog)
Agora, vamos planejar o crossmatching utilizando o LSDB, mas ainda não iremos executá-lo. Lembrando, novamente, que há uma diferença entre qual é o catálogo "direito" e qual é o "esquerdo" no crossmatching, como dito na seção anterior quando elaborarmos o cache de margem para o 2dFLenS. No nosso caso, o DES DR2 será o nosso catálogo "esquerdo", e o 2dFLenS o nosso catálogo "direito". Assim, usamos o método crossmatch sobre o catálogo do DES e passamos, como argumento, o catálogo do 2dFLenS. Os demais argumentos são o raio de busca (radius_arcsec), em arcsec, o número de objetos vizinhos (n_neighbors) que serão encontrados no catálogo da direita para cada objeto no catálogo da esquerda (o padrão é apenas um objeto vizinho, ou seja, o objeto do catálogo direito mais próximo ao objeto em questão no catálogo esquerdo) e os sufixos (suffixes) que serão usados para diferenciar os dados de ambos os catálogos nos resultados do crossmatching.
Uma observação importante é que o raio do crossmatching (radius_arcsec) não pode ser maior que o margin_threshold do cache de margem do catálogo direito, senão o lsdb exibe um erro:
ValueError: Cross match radius is greater than margin threshold.
xmatched = des_catalog.crossmatch(
_2dflens_catalog,
# Up to 1 arcsec distance, it is the default
radius_arcsec=2.0,
# Single closest object, it is the default
n_neighbors=1,
# Default would be to use names of the HATS catalogs
suffixes=("_des", "_2dflens"),
)
display(des_catalog)
display(_2dflens_catalog)
display(xmatched)
| coadd_object_id | ra | dec | mag_auto_g_dered | mag_auto_r_dered | mag_auto_i_dered | mag_auto_z_dered | mag_auto_y_dered | magerr_auto_g | magerr_auto_r | magerr_auto_i | magerr_auto_z | magerr_auto_y | flags_g | flags_r | flags_i | flags_z | flags_y | extended_class_coadd | Norder | Dir | Npix | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| npartitions=55 | ||||||||||||||||||||||
| 1157706579210928128 | int64[pyarrow] | double[pyarrow] | double[pyarrow] | double[pyarrow] | double[pyarrow] | double[pyarrow] | double[pyarrow] | double[pyarrow] | double[pyarrow] | double[pyarrow] | double[pyarrow] | double[pyarrow] | double[pyarrow] | int64[pyarrow] | int64[pyarrow] | int64[pyarrow] | int64[pyarrow] | int64[pyarrow] | int64[pyarrow] | uint8[pyarrow] | uint64[pyarrow] | uint64[pyarrow] |
| 1157776947955105792 | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 2503719917841285120 | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 2504001392817995776 | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| index | ra | dec | z | err_z | flag_des | survey | flag_survey | id_spec | Norder | Dir | Npix | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| npartitions=86 | ||||||||||||
| 1154047404513689600 | int64[pyarrow] | double[pyarrow] | double[pyarrow] | double[pyarrow] | double[pyarrow] | int64[pyarrow] | string[pyarrow] | double[pyarrow] | int64[pyarrow] | uint8[pyarrow] | uint64[pyarrow] | uint64[pyarrow] |
| 1155173304420532224 | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 2558044588346441728 | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 2576058986855923712 | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| coadd_object_id_des | ra_des | dec_des | mag_auto_g_dered_des | mag_auto_r_dered_des | mag_auto_i_dered_des | mag_auto_z_dered_des | mag_auto_y_dered_des | magerr_auto_g_des | magerr_auto_r_des | magerr_auto_i_des | magerr_auto_z_des | magerr_auto_y_des | flags_g_des | flags_r_des | flags_i_des | flags_z_des | flags_y_des | extended_class_coadd_des | Norder_des | Dir_des | Npix_des | index_2dflens | ra_2dflens | dec_2dflens | z_2dflens | err_z_2dflens | flag_des_2dflens | survey_2dflens | flag_survey_2dflens | id_spec_2dflens | Norder_2dflens | Dir_2dflens | Npix_2dflens | _dist_arcsec | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| npartitions=54 | |||||||||||||||||||||||||||||||||||
| 1157706579210928128 | int64[pyarrow] | double[pyarrow] | double[pyarrow] | double[pyarrow] | double[pyarrow] | double[pyarrow] | double[pyarrow] | double[pyarrow] | double[pyarrow] | double[pyarrow] | double[pyarrow] | double[pyarrow] | double[pyarrow] | int64[pyarrow] | int64[pyarrow] | int64[pyarrow] | int64[pyarrow] | int64[pyarrow] | int64[pyarrow] | uint8[pyarrow] | uint64[pyarrow] | uint64[pyarrow] | int64[pyarrow] | double[pyarrow] | double[pyarrow] | double[pyarrow] | double[pyarrow] | int64[pyarrow] | string[pyarrow] | double[pyarrow] | int64[pyarrow] | uint8[pyarrow] | uint64[pyarrow] | uint64[pyarrow] | double[pyarrow] |
| 1157776947955105792 | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 2503719917841285120 | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 2504001392817995776 | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
Agora, vamos executar o pipeline do crossmatching utilizando o cliente Dask (DESCOMENTE A LINHA PARA RODAR O PIPELINE).
# Run the pipeline with Dask client, it will take a while
#xmatched.to_hats(OUTPUT_HATS_DIR)
A seguir, temos as primeiras linhas da tabela contendo os dados do crossmatching.
# Look into the data
xmatched_from_disk = lsdb.read_hats(OUTPUT_HATS_DIR)
xmatched_from_disk.head()
| coadd_object_id_des | ra_des | dec_des | mag_auto_g_dered_des | mag_auto_r_dered_des | mag_auto_i_dered_des | mag_auto_z_dered_des | mag_auto_y_dered_des | magerr_auto_g_des | magerr_auto_r_des | ... | z_2dflens | err_z_2dflens | flag_des_2dflens | survey_2dflens | flag_survey_2dflens | id_spec_2dflens | Norder_2dflens | Dir_2dflens | Npix_2dflens | _dist_arcsec | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| _healpix_29 | |||||||||||||||||||||
| 1157791782152443949 | 1073555779 | 7.872479 | -32.938633 | 18.057465 | 17.141499 | 16.758852 | 16.495726 | 16.378717 | 0.0024 | 0.001319 | ... | 0.10881 | 99.0 | 4 | 2dFLenS | 4.0 | 9999 | 5 | 0 | 4113 | 0.357169 |
| 1157791859238008261 | 1073560288 | 7.778464 | -32.978252 | 21.760145 | 19.908009 | 19.160336 | 18.790243 | 18.651714 | 0.034711 | 0.007971 | ... | 0.51039 | 99.0 | 3 | 2dFLenS | 3.0 | 9999 | 5 | 0 | 4113 | 0.236634 |
| 1157793007663765486 | 1073559488 | 7.613812 | -32.971449 | 21.434824 | 20.16061 | 19.415466 | 19.052874 | 18.914614 | 0.019588 | 0.007616 | ... | 0.64152 | 99.0 | 3 | 2dFLenS | 3.0 | 9999 | 5 | 0 | 4113 | 0.438652 |
| 1157798086928282182 | 1079190881 | 8.080433 | -32.906157 | 18.973701 | 18.444038 | 18.179598 | 18.057798 | 17.961157 | 0.003494 | 0.002671 | ... | 0.10656 | 99.0 | 4 | 2dFLenS | 4.0 | 9999 | 5 | 0 | 4113 | 0.280111 |
| 1157799910512670535 | 1079184736 | 8.235737 | -32.85318 | 20.6238 | 19.086035 | 18.597113 | 18.275724 | 18.243652 | 0.01556 | 0.00489 | ... | 0.33888 | 99.0 | 4 | 2dFLenS | 4.0 | 9999 | 5 | 0 | 4113 | 0.268651 |
5 rows × 35 columns
Podemos obter também a estatística básica dos dados da tabela.
df = xmatched_from_disk.compute()
df.describe()
| coadd_object_id_des | ra_des | dec_des | mag_auto_g_dered_des | mag_auto_r_dered_des | mag_auto_i_dered_des | mag_auto_z_dered_des | mag_auto_y_dered_des | magerr_auto_g_des | magerr_auto_r_des | ... | dec_2dflens | z_2dflens | err_z_2dflens | flag_des_2dflens | flag_survey_2dflens | id_spec_2dflens | Norder_2dflens | Dir_2dflens | Npix_2dflens | _dist_arcsec | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| count | 764.0 | 764.0 | 764.0 | 764.0 | 764.0 | 764.0 | 764.0 | 764.0 | 764.0 | 764.0 | ... | 764.0 | 764.0 | 764.0 | 764.0 | 764.0 | 764.0 | 764.0 | 764.0 | 764.0 | 764.0 |
| mean | 1079214861.396597 | 8.459618 | -31.527966 | 19.814559 | 18.705034 | 18.231998 | 17.958521 | 17.86631 | 0.014016 | 0.004687 | ... | -31.527944 | 0.287005 | 99.0 | 3.841623 | 3.841623 | 9999.0 | 4.993455 | 0.0 | 4878.899215 | 0.251573 |
| std | 5793412.318042 | 0.865221 | 0.855726 | 1.400083 | 1.142922 | 1.01595 | 0.996575 | 1.007806 | 0.020505 | 0.004561 | ... | 0.85572 | 0.218182 | 0.0 | 0.365333 | 0.365333 | 0.0 | 0.080686 | 0.0 | 1775.067373 | 0.23278 |
| min | 1068874783.0 | 7.001043 | -32.998752 | 16.58913 | 15.784135 | 15.372858 | 15.087356 | 14.954291 | 0.000834 | 0.000498 | ... | -32.998768 | 0.00472 | 99.0 | 3.0 | 3.0 | 9999.0 | 4.0 | 0.0 | 2222.0 | 0.012964 |
| 25% | 1074788699.25 | 7.747955 | -32.302962 | 18.875767 | 17.982236 | 17.592261 | 17.339437 | 17.244362 | 0.003544 | 0.002016 | ... | -32.302932 | 0.141155 | 99.0 | 4.0 | 4.0 | 9999.0 | 5.0 | 0.0 | 4116.0 | 0.139627 |
| 50% | 1080049257.0 | 8.389565 | -31.583696 | 19.663832 | 18.695701 | 18.310938 | 18.05775 | 17.967001 | 0.006104 | 0.003253 | ... | -31.583644 | 0.22666 | 99.0 | 4.0 | 4.0 | 9999.0 | 5.0 | 0.0 | 4116.0 | 0.204009 |
| 75% | 1085110211.5 | 9.232875 | -30.755467 | 20.502199 | 19.325318 | 18.891093 | 18.640146 | 18.570348 | 0.012348 | 0.004894 | ... | -30.755428 | 0.338873 | 99.0 | 4.0 | 4.0 | 9999.0 | 5.0 | 0.0 | 4118.0 | 0.291419 |
| max | 1091095780.0 | 9.997029 | -30.000657 | 23.731585 | 21.639402 | 20.91231 | 20.764465 | 20.736324 | 0.171217 | 0.030343 | ... | -30.00062 | 2.24094 | 99.0 | 4.0 | 4.0 | 9999.0 | 5.0 | 0.0 | 8894.0 | 1.996786 |
8 rows × 34 columns
Análise dos resultados do X-matching¶
A seguir, vamos selecionar uma região do céu delimitada por:
print(f"R.A. min: {ra_min}")
print(f"R.A. max: {ra_max}")
print(f"DEC min: {dec_min}")
print(f"DEC max: {dec_max}")
R.A. min: 7 R.A. max: 10 DEC min: -33 DEC max: -30
Para isso, podemos usar o método polygon_search do LSDB para selecionar essa região de interesse nos catálogos, e depois executar o cálculo com o .compute().
polygon_coords = [[ra_min, dec_max], [ra_max, dec_max], [ra_max, dec_min], [ra_min, dec_min]]
des_box = des_catalog.polygon_search(polygon_coords).compute()
_2dflens_box = _2dflens_catalog.polygon_search(polygon_coords).compute()
xmatch_box = xmatched.polygon_search(polygon_coords).compute()
Convertemos, então, as coordenadas R.A. para o intervalo $(-180^{\circ}, 180^{\circ}]$.
ra_des = np.where(des_box["ra"] > 180, des_box["ra"] - 360, des_box["ra"])
ra_2dflens = np.where(_2dflens_box["ra"] > 180, _2dflens_box["ra"] - 360, _2dflens_box["ra"])
ra_x_2dflens = np.where(xmatch_box["ra_2dflens"] > 180, xmatch_box["ra_2dflens"] - 360, xmatch_box["ra_2dflens"])
Finalmente, montamos o plot. Nesse plot, os dados do DES são representados por pontos azuis, os dados do 2dFLenS por pontos verdes e os objetos do 2dFLenS que obtiveram uma correspondência a algum objeto do DES no crossmatching estão marcados em vermelho.
plt.figure(figsize=(6, 6))
plt.scatter(ra_des, des_box["dec"], s=2, alpha=0.01, marker="+", color="blue", label="DES")
plt.scatter(ra_2dflens, _2dflens_box["dec"], s=50, alpha=0.7, color="green", label="2dflens")
plt.scatter(ra_x_2dflens, xmatch_box["dec_2dflens"], s=10, alpha=0.8, color="red", label="x-matched")
plt.xlabel("R.A. (deg)")
plt.ylabel("DEC (deg)")
plt.legend(loc='lower right')
<matplotlib.legend.Legend at 0x7ff216160990>
Podemos fazer também um gráfico com o Holoviews exibindo apenas os objetos do DES que obtiveram uma correspondência a algum objeto do 2dFLenS no crossmatching. O interessante desse gráfico é que podemos interagir com ele, utilizando, por exemplo, a ferramenta Hover para obter as coordenadas R.A. e DEC de um determinado objeto e a distância dele ao objeto do 2dFLenS com o qual ele foi associado no crossmatching. Adicionamos, ainda, uma barra de cor que indica a distância dos objetos do DES ao seu correpondente do 2dFLenS, de acordo com o crossmatching.
# Criar o gráfico 2D com Holoviews
points = hv.Points(df, kdims=['ra_des', 'dec_des'], vdims=['_dist_arcsec'])
# Configurar a coloração e ajustar o tamanho dos labels dos eixos
points = points.opts(
width=750, height=500,
color='_dist_arcsec', cmap='Viridis', colorbar=False, tools=['hover'],
size=8,
xlabel='RA (deg)', ylabel='DEC (deg)',
fontsize={'xticks': 12, 'yticks': 12, 'xlabel': 14, 'ylabel': 14}
)
# Personalizar o ColorBar (do Bokeh)
color_mapper = LinearColorMapper(palette=Viridis256, low=df['_dist_arcsec'].min(), high=df['_dist_arcsec'].max())
color_bar = ColorBar(color_mapper=color_mapper, label_standoff=12, location=(0,0), title='Dist. (Arcsec)')
color_bar.title_text_font_size = '14pt'
color_bar.major_label_text_font_size = '12pt'
# Criar layout Holoviews com o gráfico e o color bar
layout = points.opts(colorbar=True, colorbar_opts={'location': 'right', 'title': 'Dist. (Arcsec)'})
# Salvar o gráfico em HTML
hv.save(layout, 'separation_scatter.html')
IFrame('separation_scatter.html', width=1200, height=600)
Podemos também checar se existem objetos de 2dFLenS que foram associados a mais de um objeto do DES, por causa do cache de margem.
df_duplicates = df[df.duplicated(subset=['ra_2dflens'])]
df_duplicates
| coadd_object_id_des | ra_des | dec_des | mag_auto_g_dered_des | mag_auto_r_dered_des | mag_auto_i_dered_des | mag_auto_z_dered_des | mag_auto_y_dered_des | magerr_auto_g_des | magerr_auto_r_des | ... | z_2dflens | err_z_2dflens | flag_des_2dflens | survey_2dflens | flag_survey_2dflens | id_spec_2dflens | Norder_2dflens | Dir_2dflens | Npix_2dflens | _dist_arcsec | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| _healpix_29 | |||||||||||||||||||||
| 1157844720795156399 | 1074784789 | 7.754276 | -32.272302 | 20.911825 | 19.519079 | 19.064787 | 18.784485 | 18.658978 | 0.007786 | 0.002557 | ... | 0.29659 | 99.0 | 4 | 2dFLenS | 4.0 | 9999 | 5 | 0 | 4113 | 1.996786 |
| 1158731662151250046 | 1075092634 | 7.593506 | -31.485583 | 20.751265 | 19.195852 | 18.647636 | 18.333942 | 18.20488 | 0.016503 | 0.004817 | ... | 0.32938 | 99.0 | 4 | 2dFLenS | 4.0 | 9999 | 5 | 0 | 4116 | 0.183559 |
| 1158815080495274174 | 1080955910 | 8.4229 | -30.640853 | 23.127935 | 21.639402 | 20.268812 | 19.729223 | 19.616301 | 0.06136 | 0.020421 | ... | 0.71906 | 99.0 | 3 | 2dFLenS | 3.0 | 9999 | 5 | 0 | 4116 | 1.648327 |
| 1158883278202374490 | 1087158439 | 9.404071 | -30.545324 | 19.993082 | 19.530603 | 19.269825 | 19.168812 | 19.127234 | 0.002478 | 0.002042 | ... | 0.17454 | 99.0 | 4 | 2dFLenS | 4.0 | 9999 | 5 | 0 | 4117 | 1.776188 |
4 rows × 35 columns
if not df_duplicates.empty:
for i in np.arange(0,len(df_duplicates),1):
id_duplicate = df_duplicates['index_2dflens'].values[i]
print(f"ID do objeto do 2dFLenS: {id_duplicate}")
print(f"ID dos objetos do DES associados a esse mesmo objeto do 2dFLenS: {df[df['index_2dflens'] == id_duplicate]['coadd_object_id_des'].tolist()}")
print('\n')
ID do objeto do 2dFLenS: 225608 ID dos objetos do DES associados a esse mesmo objeto do 2dFLenS: [1074784520, 1074784789] ID do objeto do 2dFLenS: 203452 ID dos objetos do DES associados a esse mesmo objeto do 2dFLenS: [1075093286, 1075092634] ID do objeto do 2dFLenS: 203515 ID dos objetos do DES associados a esse mesmo objeto do 2dFLenS: [1080955650, 1080955910] ID do objeto do 2dFLenS: 245476 ID dos objetos do DES associados a esse mesmo objeto do 2dFLenS: [1087157360, 1087158439]
Além disso, podemos fazer também o histograma das distâncias de separação dos objetos do crossmatching.
# Convertendo a coluna _dist_arcsec para um array numpy.
df['_dist_arcsec'] = df['_dist_arcsec'].to_numpy()
# Criar um Dataset a partir do DataFrame
dataset = hv.Dataset(df, kdims='_dist_arcsec')
# Aplicar a operação de histograma
hist = hv.operation.histogram(dataset, dimension='_dist_arcsec', normed=False, bins=20)
# Personalizar o histograma
hist.opts(
xlabel='Distância (arcsec)',
ylabel='Frequência',
title='Histograma das distâncias de separação',
color='blue',
tools=['hover'],
width=750,
height=500
)
# Mostrar o histograma
hv.save(hist, 'separation_histogram.html')
IFrame('separation_histogram.html', width=1200, height=600)